오카자키 절편
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
오카자키 절편은 1960년대 오카자키 레이지와 오카자키 쓰네코 부부에 의해 발견된, DNA 복제 과정에서 생성되는 짧은 DNA 조각이다. 기존에는 DNA 복제가 연속적으로 일어난다고 알려져 있었으나, 오카자키 부부는 대장균을 이용한 실험을 통해 DNA 중합효소가 5'에서 3' 방향으로만 DNA를 합성하며, 지연 가닥에서는 짧은 DNA 조각들이 먼저 합성된 후 연결된다는 것을 밝혀냈다. 오카자키 절편은 DNA 복제 과정에서 RNA 프라이머 제거 및 DNA 연결 효소에 의해 결합되어 새로운 DNA 가닥을 형성하는 데 중요한 역할을 하며, 유전체 안정성 유지에 기여한다. 오카자키 절편의 생성 및 처리 과정은 다양한 효소에 의해 조절되며, 이러한 과정의 결함은 유전 질환 및 암 발생과 연관될 수 있다.
DNA가 복제될 때, 이중 나선이 풀리고 DNA 헬리케이스에 의해 상보 가닥이 분리되면 DNA 프라이머제와 DNA 중합 효소가 작용하여 새로운 상보 가닥을 생성한다. DNA 중합 효소에 의한 DNA 합성은 5'에서 3' 방향으로만 진행될 수 있지만, 지연 가닥에서는 3'에서 5' 방향으로의 합성이 필요하다.[28] 따라서 DNA 합성은 단계적으로 진행된다. 먼저 DNA 프라이머제에 의해 몇 개의 염기로 구성된 짧은 RNA (프라이머)이 합성되고, 이어서 3' 말단에서 DNA 중합 효소 III에 의해 DNA가 합성된다(단, 진핵생물은 DNA 중합 효소 δ, 크렌 고세균은 DNA 중합 효소 B, 유리 고세균은 DNA 중합 효소 D를 사용한다). 이렇게 생성되는 것이 오카자키 절편이다. 오카자키 절편의 길이는 진정 세균에서는 1000 - 2000 염기 정도, 진핵생물 및 고세균에서는 100 - 200 염기 정도이다.
2. 역사
기요코 사카베, 오카자키 레이지, 오카자키 쓰네코는 DNA 복제가 불연속적인 과정이라는 가설을 뒷받침하는 실험적 증거를 제공했다. 이전에는 복제가 3'에서 5' 방향과 5'에서 3' 방향 모두에서 연속적으로 일어난다고 일반적으로 받아들여졌다. 3'와 5'는 핵산의 데옥시리보스 고리에서 특정 번호를 매긴 탄소이며, 가닥의 방향성을 나타낸다. 1967년, 오카자키 쓰네코와 오가와 토루는 DNA 중합효소가 5'에서 3' 방향으로만 복제되며, 3'에서 5' 방향으로의 연속적인 복제를 보여주는 메커니즘은 없다고 주장했다.[5]
2. 1. 발견
오카자키 레이지와 오카자키 쓰네코 부부는 1960년대에 일련의 실험들을 통해 오카자키 절편을 발견하였다. 이들은 대장균(''Escherichia coli'')을 이용한 실험에서 DNA 복제가 불연속적인 방식으로 일어난다는 증거를 제시했다.[5][6][7]
연구팀은 불연속적인 복제가 사용된다면, 복제 지점에서 합성된 짧은 DNA 가닥이 오래된 가닥에 5'에서 3' 방향으로 부착될 수 있다고 가설을 세웠다.[5]
DNA가 사용하는 복제 방식을 실험적으로 확인하기 위해, 연구팀은 새로 복제된 ''대장균'' 염색체 영역에 펄스 표지를 하고, DNA를 변성시킨 후 추출했다. 다수의 방사성 단편은 복제 방식이 불연속적일 가능성이 높다는 것을 의미했다. 이 가설은 짧은 DNA 가닥을 연결하는 효소인 리가아제의 발견으로 더욱 뒷받침되었다.[6]
1968년, 레이지와 오카자키 쓰네코는 새롭게 합성된 DNA 가닥에 대한 추가적인 증거를 수집했다. 그들은 "리가아제의 기능이 일시적으로 손상된 조건에서 새로 합성된 짧은 DNA 사슬이 세포에 축적될 것"이라고 가설을 세웠다. 온도가 민감한 폴리뉴클레오티드 리가아제를 생성하는 박테리오파지 T4에 ''대장균''을 감염시켰다. T4 파지에 감염된 세포는 고온에 노출되었을 때, 가설에서 예측한 대로 다수의 짧고 새롭게 합성된 DNA 사슬을 축적했다. 이 실험은 불연속적 복제와 폴리뉴클레오티드 리가아제에 의한 연결에 대한 오카자키의 가설을 더욱 뒷받침했다. 또한, 이 실험은 추출 과정에서 짧은 사슬이 생성된다는 개념을 반증했다.[7]
오카자키 부부는 대장균 체내에서 DNA 복제를 수행하는 실험을 진행했다. 우선, 대장균에 방사성 티미딘을 섭취하게 했다(펄스 표지).[27][29] DNA 복제는 하나의 세포 내에서 1,000 뉴클레오티드/초의 속도로 진행되기 때문에, 저온으로 반응 속도를 늦추고, 표지 시간을 단축하여 방사성 티미딘을 관찰했다.[27] 그 결과, 1,000 - 2,000 염기쌍의 짧은 DNA를 추출했다. 이는 지연 가닥에서 불연속 복제가 이루어지고 있을 가능성을 의미했다.
이후, 미국 체류 중에 대장균과 T4 파지 내에서 DNA 연결 효소가 발견되었으며, DNA 연결 효소가 작용하는 조건과 작용하지 않는 조건에서 표지 실험을 진행하여, DNA 연결 효소가 작용하지 않는 조건에서는 장쇄 형성이 억제되고 단쇄가 축적되는 것을 확인하여 가설을 더욱 뒷받침했다.[27]
2. 2. 초기 가설과 실험적 증거
기요코 사카베, 오카자키 레이지 그리고 오카자키 쓰네코의 연구는 DNA 복제가 불연속적인 과정이라는 가설을 뒷받침하는 실험적 증거를 제공했다. 이전에는 복제가 3'에서 5' 방향과 5'에서 3' 방향 모두에서 연속적으로 일어난다고 일반적으로 받아들여졌다. 3'와 5'는 핵산의 데옥시리보스 고리에서 특정 번호를 매긴 탄소이며, 가닥의 방향성을 나타낸다. 1967년, 오카자키 쓰네코와 오가와 토루는 DNA 중합효소가 5'에서 3' 방향으로만 복제되며, 3'에서 5' 방향으로의 연속적인 복제를 보여주는 메커니즘은 없다고 주장했다. 연구팀은 불연속적인 복제가 사용된다면, 복제 지점에서 합성된 짧은 DNA 가닥이 오래된 가닥에 5'에서 3' 방향으로 부착될 수 있다고 가설을 세웠다.[5]
DNA 복제 방식을 실험적으로 확인하기 위해, 연구팀은 새로 복제된 ''대장균'' 염색체 영역에 펄스 표지를 하고, DNA를 변성시킨 후 추출했다. 다수의 방사성 단편은 복제 방식이 불연속적일 가능성이 높다는 것을 의미했다. 이 가설은 짧은 DNA 가닥을 연결하는 효소인 리가아제의 발견으로 더욱 뒷받침되었다.[6]
1968년, 레이지와 오카자키 쓰네코는 새롭게 합성된 DNA 가닥에 대한 추가적인 증거를 수집했다. 그들은 "리가아제의 기능이 일시적으로 손상된 조건에서 새로 합성된 짧은 DNA 사슬이 세포에 축적될 것"이라고 가설을 세웠다. 온도가 민감한 폴리뉴클레오티드 리가아제를 생성하는 박테리오파지 T4에 ''대장균''을 감염시켰다. T4 파지에 감염된 세포는 고온에 노출되었을 때, 가설에서 예측한 대로 다수의 짧고 새롭게 합성된 DNA 사슬을 축적했다. 이 실험은 불연속적 복제와 폴리뉴클레오티드 리가아제에 의한 연결에 대한 오카자키의 가설을 더욱 뒷받침했다. 또한, 이 실험은 추출 과정에서 짧은 사슬이 생성된다는 개념을 반증했다.[7]
오카자키의 실험은 DNA의 복제 과정과 나중에 오카자키 절편으로 알려진 짧고 새롭게 합성된 DNA 사슬의 존재에 대한 광범위한 정보를 제공했다.
오카자키 겐지와 오카자키 쓰네코는 대장균 체내에서 DNA 복제를 수행하는 실험을 진행했다. 우선, 대장균에 방사성 티미딘을 섭취하게 했다(펄스 표지).[27][29] 하지만 DNA 복제는 하나의 세포 내에서 1,000 뉴클레오티드/초의 속도로 진행되기 때문에, 저온으로 반응 속도를 늦추고, 표지 시간을 단축하여 방사성 티미딘을 관찰했다.[27] 그 결과, 1,000 - 2,000 염기쌍의 짧은 DNA를 추출했다. 이는 지연 가닥에서 불연속 복제가 이루어지고 있을 가능성을 의미했다.
이후, 미국 체류 중에 대장균과 T4 파지 내에서 DNA 연결 효소가 발견되었으며, DNA 연결 효소가 작용하는 조건과 작용하지 않는 조건에서 표지 실험을 진행하여, DNA 연결 효소가 작용하지 않는 조건에서는 장쇄 형성이 억제되고 단쇄가 축적되는 것을 확인하여 가설을 더욱 뒷받침했다.[27]
2. 3. 추가 증거와 메커니즘 규명
DNA 리가아제(DNA ligase)의 발견은 오카자키 절편들이 서로 연결되어 긴 DNA 가닥을 형성한다는 것을 설명하는 데 중요한 역할을 했다. 오카자키 부부는 DNA 리가아제의 기능을 억제한 조건에서 짧은 DNA 조각들이 축적되는 것을 확인함으로써, 이들의 가설을 더욱 뒷받침했다.[7][27]
1968년, 레이지와 오카자키 쓰네코는 새롭게 합성된 DNA 가닥에 대한 추가적인 증거를 수집했다. 그들은 짧은 DNA 사슬이 폴리뉴클레오티드 리가아제에 의해 연결되는 불연속적 복제가 DNA 합성에 사용되는 메커니즘이라면, "리가아제의 기능이 일시적으로 손상된 조건에서 새로 합성된 짧은 DNA 사슬이 세포에 축적될 것"이라고 가설을 세웠다. 온도가 민감한 폴리뉴클레오티드 리가아제를 생성하는 박테리오파지 T4에 ''대장균''을 감염시켰다. T4 파지에 감염된 세포는 고온에 노출되었을 때, 가설에서 예측한 대로 다수의 짧고 새롭게 합성된 DNA 사슬을 축적했다. 이 실험은 불연속적 복제와 폴리뉴클레오티드 리가아제에 의한 연결에 대한 오카자키의 가설을 더욱 뒷받침했다. 또한, 이 실험은 추출 과정에서 짧은 사슬이 생성된다는 개념을 반증했다.[7]
오카자키 겐지와 오카자키 쓰네코는 대장균 체내에서 DNA 복제를 수행하는 실험을 진행했다. 우선, 대장균에 방사성 티미딘을 섭취하게 했다(펄스 표지)[27][29]。 DNA 복제는 하나의 세포 내에서 1,000 뉴클레오티드/초의 속도로 진행되기 때문에, 저온으로 반응 속도를 늦추고, 표지 시간을 단축하여 방사성 티미딘을 관찰했다[27]。 그 결과, 1,000 - 2,000 염기쌍의 짧은 DNA를 추출했다. 이는 지연 가닥에서 불연속 복제가 이루어지고 있을 가능성을 의미했다.
이후, 미국 체류 중에 대장균과 T4 파지 내에서 DNA 연결 효소가 발견되었으며, DNA 연결 효소가 작용하는 조건과 작용하지 않는 조건에서 표지 실험을 진행하여, DNA 연결 효소가 작용하지 않는 조건에서는 장쇄 형성이 억제되고 단쇄가 축적되는 것을 확인하여 가설을 더욱 뒷받침했다[27]。
3. 기작 (Pathways)
이후 RNA 부분은 RNase H에 의해 분해되고 DNA 중합 효소 I에 의해 분해 및 DNA 재합성이 이루어진다. 조각 사이에 남은 닉 (포스포디에스터 결합이 끊어진 부분)은 DNA 연결 효소에 의해 결합된다.
3. 1. 짧은 플랩 경로 (Short Flap Pathway)
진핵생물의 짧은 플랩 경로에서 DNA의 지연 가닥은 짧은 간격으로 시동된다. 짧은 경로에서만 FEN1 핵산 분해 효소가 관여한다. DNA 중합효소 δ (Pol δ)는 종종 하류의 시동된 오카자키 절편과 마주치고 RNA/DNA 개시 프라이머를 5' 플랩으로 밀어낸다.[8] FEN1 5'-3' 엔도뉴클레아제는 5' 플랩이 밀려났음을 인식하고 절단하여 연결을 위한 기질을 생성한다. 이 방법으로 Pol α에 의해 합성된 프라이머가 제거된다. DNA 연결 효소는 FEN1에 의해 만들어진 틈(nick)을 봉합하고 기능적인 연속적인 이중 가닥 DNA를 생성한다. PCNA는 FEN1과 DNA 연결 효소 모두에 대해 단백질의 효소 기능을 시뮬레이션한다. 이러한 상호 작용은 지연 DNA 가닥의 적절한 연결을 생성하는 데 매우 중요하다. Pol δ와 FEN1에 의한 순차적인 가닥 치환 및 절단은 연결 전에 전체 개시 RNA를 제거하는 데 도움이 된다. 많은 변위가 일어나야 하고 개시 프라이머를 제거하기 위해 절단 반응이 필요하다. 생성되고 처리된 플랩은 짧은 플랩 경로에 의해 성숙된다.
3. 2. 긴 플랩 경로 (Long Flap Pathway)
FEN1의 활성이 일시적으로 억제되거나, DNA 중합효소 δ에 의해 밀려난 플랩이 길어질 경우에 일어나는 경로이다. RPA(Replication protein A)가 긴 플랩에 결합하여 FEN1에 의한 절단을 막는다. DNA2가 긴 플랩을 절단하여 FEN1이 작용할 수 있는 기질을 만든다. 이후 FEN1과 DNA 리가아제 I에 의해 RNA 프라이머 제거 및 DNA 연결이 일어난다.[1] DNA2는 뉴클레아제 활성을 위해 FEN1의 역할을 대신 할 수 있지만, 효율적인 과정은 아니다.[1]
3. 3. 대체 경로 (Alternate Pathway)
최근까지 오카자키 절편을 처리하는 경로는 두 가지로 알려져 있었다. 그러나 현재 연구에 따르면 오카자키 절편화 및 DNA 복제에 대한 새로운 경로가 존재한다는 결론이 나왔다. 이 대체 경로는 Pol δ와 Pif1 효소를 포함하며, Pol δ와 FEN1이 수행하는 것과 동일한 플랩 제거 과정을 수행한다.[9]
4. 관여하는 효소 (Enzymes involved in fragment formation)
DNA 복제 과정에서 DNA는 헬리케이스에 의해 이중 나선이 풀리고, DNA 프라이머제와 DNA 중합 효소가 새로운 상보 가닥을 생성한다. DNA 중합 효소는 5'에서 3' 방향으로만 DNA를 합성할 수 있지만, 지연 가닥에서는 3'에서 5' 방향으로의 합성이 필요하다.[28] 따라서 DNA 합성은 단계적으로 진행된다.
먼저 DNA 프라이머제에 의해 몇 개의 염기로 구성된 짧은 RNA 조각인 프라이머가 합성되고, 이 3' 말단에서 DNA 중합 효소 III (진핵생물은 DNA 중합 효소 δ, 크렌 고세균은 DNA 중합 효소 B, 유리 고세균은 DNA 중합 효소 D)에 의해 DNA가 합성된다. 이렇게 만들어진 짧은 DNA 조각이 오카자키 절편이다. 진정 세균에서 오카자키 절편은 1000 - 2000 염기, 진핵생물 및 고세균에서는 100 - 200 염기 정도의 길이를 가진다.
이후 RNA 부분은 RNase H에 의해 분해되고, DNA 중합 효소 I에 의해 DNA가 다시 합성된다. 조각 사이에 남은 닉 (포스포디에스터 결합이 끊어진 부분)은 DNA 연결 효소에 의해 연결된다.
4. 1. 프리마아제 (Primase)
프리마아제는 RNA 프라이머를 지연 가닥에 추가하여, 오카자키 절편을 5'에서 3' 방향으로 합성할 수 있게 한다. 하지만 프리마아제는 DNA 중합효소가 선도 가닥에서 DNA를 합성하는 속도보다 훨씬 느린 속도로 RNA 프라이머를 생성한다. 지연 가닥의 DNA 중합효소 또한 RNA 프라이머를 따라 오카자키 절편을 만들기 위해 지속적으로 재활용되어야 한다. 이것은 지연 가닥의 합성 속도를 선도 가닥보다 훨씬 느리게 만든다. 이를 해결하기 위해 프리마아제는 일시적인 정지 신호로 작용하여, DNA 복제 과정에서 복제 분기점의 진행을 잠시 멈춘다. 이 분자 과정은 선도 가닥이 지연 가닥을 앞지르는 것을 방지한다.[10]DNA가 복제될 때 이중 나선이 풀리고 상보 가닥이 DNA 헬리케이스에 의해 분리되면 DNA 프라이머제와 DNA 중합 효소가 작용하여 새로운 상보 가닥을 생성한다. DNA 중합 효소에 의한 DNA 합성은 5'에서 3' 방향으로만 진행될 수 있지만, 지연 가닥에서는 3'에서 5' 방향으로의 합성이 필요하다.[28] 따라서 DNA 합성은 단계적으로 진행된다. 먼저 DNA 프라이머제에 의해 몇 개의 염기로 구성된 짧은 RNA (프라이머)이 합성되고, 이어서 3' 말단에서 DNA 중합 효소 III에 의해 DNA가 합성된다(단, 진핵생물은 DNA 중합 효소 δ, 크렌 고세균은 DNA 중합 효소 B, 유리 고세균은 DNA 중합 효소 D를 사용한다).
4. 2. DNA 중합효소 δ (DNA polymerase δ)
DNA 중합효소 δ는 지연 가닥에서 오카자키 절편을 합성하는 주요 효소이다. 5'→3' 방향으로 DNA를 합성하며, 이전 오카자키 절편의 RNA 프라이머를 밀어내는 역할을 한다.[28] 진핵생물은 DNA 중합효소 δ를 사용한다.DNA 중합효소 δ는 홀로효소 형태로 들어가 합성이 시작된다. 합성 과정은 이전 오카자키 절편의 5' 말단에 도달할 때까지 계속되며, 오카자키 절편 처리는 새로 합성된 절편을 지연 가닥에 연결하는 과정으로 진행된다.
4. 3. DNA 리가아제 I (DNA ligase I)
지연 가닥 합성 과정에서, DNA 리가아제 I은 DNA 중합효소 δ가 RNA 프라이머를 DNA 뉴클레오타이드로 대체한 후 오카자키 절편을 연결하는 역할을 한다.[11] 연결되지 않은 오카자키 절편은 DNA를 절단하는 이중 가닥 절단을 유발할 수 있는데, 이러한 절단이 충분히 복구되지 않으면 세포에 치명적일 수 있다.[11]증식 세포 핵 항원(PCNA)은 DNA 리가아제 I이 오카자키 절편을 연결하는 기능을 돕는다.[12] DNA 리가아제 I의 PCNA 결합 부위가 비활성화되면 오카자키 절편을 연결하는 능력이 크게 떨어진다.[12][13][14] 따라서 제안된 메커니즘은 다음과 같다. PCNA-DNA 중합효소 δ 복합체가 오카자키 절편을 합성한 후 DNA 중합효소 δ가 방출되고, DNA 리가아제 I이 PCNA에 결합하여 지연 가닥의 절단 부위에 고정되어 포스포다이에스터 결합 형성을 촉매한다.[12][13][14]
진핵생물에서 오카자키 절편의 합성은 DNA 프라이머제가 합성한 짧은 RNA 프라이머의 3' 말단에서 DNA 중합 효소 δ가 DNA를 합성하면서 시작된다. 이후 RNA 부분은 RNase H에 의해 분해되고, DNA 중합 효소 I에 의해 DNA가 다시 합성된다. 조각 사이에 남은 닉 (포스포다이에스터 결합이 끊어진 부분)은 DNA 연결 효소에 의해 연결된다.
4. 4. 플랩 엔도뉴클레아제 1 (Flap endonuclease 1, FEN1)
플랩 엔도뉴클레아제 1 (FEN1)은 오카자키 절편 처리 과정에서 DNA 중합효소와 함께 작용하여 RNA 프라이머를 제거한다. DNA 중합효소가 지연 가닥 합성 과정에서 가닥을 밀어낼 때 5' 리보뉴클레오티드와 5' 플랩을 제거하며, 이는 닉 번역 과정을 통해 연결을 위한 닉을 생성한다. FEN1의 기능은 오카자키 절편 성숙에 필수적이며, DNA 염기 복구 과정에서 손상된 뉴클레오티드가 플랩으로 밀려날 때도 FEN1에 의해 제거된다.[28]4. 5. Dna2 엔도뉴클레아제 (Dna2 endonuclease)
Dna2 엔도뉴클레아제는 특정한 구조를 가지고 있지 않으며, 그 특성이 잘 알려져 있지 않지만, 자유 말단(ssDNA)을 가진 단일 가닥 DNA로 언급될 수 있다. Dna2 엔도뉴클레아제는 오카자키 절편 과정 동안 FEN1을 남기는 긴 DNA 플랩을 절단하는 데 필수적이다.[17][18][19][20][21] Dna2 엔도뉴클레아제는 오카자키 절편의 개시 RNA 세그먼트 제거를 담당하며, 다양한 DNA 대사 과정에서 생성되는 중간체에서 중추적인 역할을 하며 텔로미어 유지에 기능한다.[17][18][19][20][21]Dna2 엔도뉴클레아제는 5' 말단에 말단 RNA 세그먼트가 부착될 때 활성화되는데, 이는 5'에서 3' 방향으로 이동하기 때문이다. 단일 가닥 DNA 결합 단백질 RPA가 존재할 때, DNA 5' 플랩이 너무 길어져서 니크가 FEN1의 기질로 더 이상 적합하지 않게 된다. 이는 FEN1이 5'-플랩을 제거하는 것을 방지한다. 따라서 Dna2의 역할은 이러한 절편의 3' 말단을 줄여 FEN1이 플랩을 절단할 수 있게 하고 오카자키 절편 성숙을 더 효율적으로 만드는 것이다. 오카자키 과정 동안 Dna2 헬리케이즈와 엔도뉴클레아제는 분리될 수 없다. Dna2 엔도뉴클레아제는 활성을 위해 5'-꼬리 포크 구조에 의존하지 않는다. 비생산적인 결합은 FEN1 절단 및 추적을 막는 것으로 알려져 있다. ATP가 활성을 감소시키지만 3'-말단 라벨의 방출을 촉진하는 것으로 알려져 있다. 연구에 따르면 Dna2 엔도뉴클레아제와 FEN1의 새로운 모델이 오카자키 절편 성숙에 부분적으로 책임이 있는 것으로 나타났다.[20][18][17][22]
5. 생물학적 기능 (Biological function)
DNA 복제 과정에서 지연 가닥에 짧게 생성되는 오카자키 절편은 DNA 합성에 필수적인 역할을 한다. 이 절편들은 DNA 연결 효소에 의해 연결되어 완전한 DNA 가닥을 형성한다. 오카자키 절편의 성숙 과정에 결함이 생기면 DNA 가닥이 끊어지거나 다양한 형태의 염색체 이상이 발생할 수 있으며, [1] 이러한 돌연변이는 생물의 외형, 염색체 수 등에 영향을 미치고, 더 나아가 유전자 풀에 결함을 일으킬 수 있다.[1]
5. 1. DNA 복제의 완성
새로 합성된 DNA, 즉 오카자키 절편은 DNA 연결 효소에 의해 결합되어 새로운 DNA 가닥을 형성한다. DNA가 합성될 때 두 가닥이 생성된다. 선도 가닥은 지속적으로 합성되며, 이 과정에서 연장되어 지연 가닥(오카자키 절편)에 사용되는 주형을 노출시킨다. DNA 복제 과정에서 DNA와 RNA 프라이머는 오카자키 절편이 결합할 수 있도록 DNA의 지연 가닥에서 제거된다. 이 과정은 매우 흔하게 발생하므로, 오카자키 절편 성숙은 DNA 복제가 한 번 완료되는 동안 약 백만 번 정도 일어난다. 오카자키 절편 성숙이 일어나려면 RNA 프라이머가 연결될 절편에 세그먼트를 생성해야 한다. 이것은 지연 가닥에서 DNA 합성을 위한 구성 요소로 사용된다. 주형 가닥에서 중합 효소는 복제 분기점과 반대 방향으로 합성된다. 주형이 불연속되면 오카자키 절편이 생성된다. 오카자키 절편 성숙의 결함은 잠재적으로 DNA의 가닥이 끊어지고 다양한 형태의 염색체 이상을 유발할 수 있다.[1] 이러한 염색체의 돌연변이는 외형, 세트 수 또는 개별 염색체 수에 영향을 미칠 수 있다. 염색체는 각 특정 종에 대해 고정되어 있기 때문에, 이는 또한 DNA를 변화시키고 해당 종의 유전자 풀에 결함을 일으킬 수 있다.[1]5. 2. 유전체 안정성 유지
오카자키 절편의 정확한 합성과 연결은 유전체의 안정성을 유지하는 데 중요하다. 새로 합성된 DNA인 오카자키 절편은 DNA 연결 효소에 의해 결합되어 새로운 DNA 가닥을 형성한다. DNA 복제 과정에서 DNA와 RNA 프라이머는 오카자키 절편이 결합할 수 있도록 DNA의 지연 가닥에서 제거된다. 이 과정은 매우 흔하게 발생하므로, 오카자키 절편 성숙은 DNA 복제가 한 번 완료되는 동안 약 백만 번 정도 일어난다. 오카자키 절편 성숙이 일어나려면 RNA 프라이머가 연결될 절편에 세그먼트를 생성해야 한다. 이것은 지연 가닥에서 DNA 합성을 위한 구성 요소로 사용된다. 주형 가닥에서 중합효소는 복제 분기점과 반대 방향으로 합성된다. 주형이 불연속적이면 오카자키 절편이 생성된다. 오카자키 절편 성숙의 결함은 잠재적으로 DNA 가닥이 끊어지고 다양한 형태의 염색체 이상을 유발할 수 있다. 이러한 염색체의 돌연변이는 외형, 세트 수 또는 개별 염색체 수에 영향을 미칠 수 있다. 염색체는 각 특정 종에 대해 고정되어 있기 때문에, 이는 또한 DNA를 변화시키고 해당 종의 유전자 풀에 결함을 일으킬 수 있다.6. 원핵생물과 진핵생물의 차이 (Differences in prokaryotes and eukaryotes)
원핵생물과 진핵생물은 모두 오카자키 절편을 갖지만, 몇 가지 중요한 차이점이 존재한다.[23]
- DNA 복제 속도 및 오카자키 절편 길이: 원핵생물은 진핵생물보다 DNA 복제 속도가 빠르며, 오카자키 절편의 길이도 더 길다.
- 텔로미어: 진핵생물은 염색체 끝부분인 텔로미어를 복제하는 특별한 방식을 가지고 있지만, 원핵생물은 환형 염색체를 가지고 있어 텔로미어가 없다.
- 세포 구조 및 DNA 양: 원핵생물 세포는 구조가 단순하고, 핵과 세포 소기관이 없으며, DNA는 단일 염색체 형태이다. 반면 진핵생물 세포는 여러 소기관을 가진 핵과 선형 염색체로 배열된 더 많은 DNA를 가지고 있다. (진핵생물 세포는 원핵생물 세포보다 약 25배 많은 DNA를 갖는다.)
DNA 복제 단계는 원핵생물과 진핵생물에서 유사하다. DNA 헬리케이스가 DNA 이중 나선을 풀고, DNA 중합효소가 새로운 DNA 가닥을 만든다. 이 과정은 반보존적 복제 패턴을 따르며, DNA의 개별 가닥은 다른 방향으로 생성되어 선도 가닥과 지연 가닥이 만들어진다. 지연 가닥은 오카자키 절편으로 합성된 후 연결된다.
6. 1. 오카자키 절편의 길이
원핵생물과 진핵생물에서 오카자키 절편의 길이는 다르다. 원핵생물은 진핵생물보다 훨씬 긴 오카자키 절편을 갖는다. 진핵생물의 오카자키 절편은 일반적으로 100~200개의 뉴클레오타이드로 구성되는 반면, 원핵생물 ''대장균''(E. coli)의 절편은 1,000~2,000개의 뉴클레오타이드로 구성될 수 있다.[23] 이러한 차이의 이유는 알려져 있지 않다.진정 세균에서 오카자키 절편의 길이는 1,000 - 2,000 염기 정도이고, 진핵생물 및 고세균에서는 100 - 200 염기 정도이다.[28]
6. 2. 복제 기원 (Origin of replication)
오카자키 절편은 원핵생물과 진핵생물 모두에 존재한다.[23] 진핵생물의 DNA 분자는 원핵생물의 환형 분자와 달리 더 크고 일반적으로 여러 복제 기원을 갖는다. 이는 각 진핵생물 염색체가 여러 복제 기원을 가진 많은 DNA 복제 단위로 구성됨을 의미한다. 이와 비교하여, 원핵생물 DNA는 단일 복제 기원만을 갖는다. 진핵생물에서 이러한 복제 분기점은 DNA를 따라 수없이 많으며 복제 중 DNA에 "버블"을 형성한다. 복제 분기점은 자율 복제 서열(ARS)이라고 하는 특정 지점에서 형성된다. 진핵생물은 클램프 로더 복합체와 증식 세포 핵 항원이라고 하는 6개의 단위 클램프를 가지고 있다.[24] 복제 분기점의 효율적인 이동은 ATP 의존적 클램프 로더 복합체에 의해 지연 DNA 가닥의 새로 프라이밍된 부위에 슬라이딩 클램프를 신속하게 배치하는 데 크게 의존한다. 이는 오카자키 절편의 조각별 생성이 선도 가닥에서 DNA의 연속적인 합성을 따라갈 수 있음을 의미한다. 이러한 클램프 로더 복합체는 모든 진핵생물의 특징이며, 원핵생물과 진핵생물에서 오카자키 절편의 합성에 존재하는 몇 가지 사소한 차이점을 구분한다.[25]오카자키 절편의 길이도 원핵생물과 진핵생물에서 다르다. 원핵생물은 진핵생물보다 훨씬 더 긴 오카자키 절편을 갖는다. 진핵생물은 일반적으로 100~200개의 뉴클레오티드로 구성된 오카자키 절편을 갖는 반면, 원핵생물 ''대장균''(E. coli)의 절편은 2,000개의 뉴클레오티드로 구성될 수 있다.
각 진핵생물 염색체는 여러 복제 기원을 가진 많은 DNA 복제 단위로 구성된다. 이와 비교하여, 원핵생물 대장균 염색체는 단일 복제 기원만을 갖는다. 원핵생물의 복제는 세포질 내부에서 발생하며, 이는 약 100~200개 이상의 뉴클레오티드로 구성된 복제를 시작한다. 진핵생물 DNA 분자는 약 50,000개 이상의 레플리콘을 상당히 많이 가지고 있지만, 모든 레플리콘에서 동시에 복제가 발생하지는 않는다. 진핵생물에서 DNA 복제는 핵에서 일어난다.
원핵생물 세포는 구조가 더 단순하며, 핵, 소기관이 없고, 단일 염색체 형태로 DNA가 거의 없다. 진핵생물 세포는 여러 소기관이 있는 핵과 선형 염색체로 배열된 더 많은 DNA를 가지고 있다. 평균적인 진핵생물 세포는 원핵생물 세포보다 약 25배 더 많은 DNA를 가지고 있다. 복제는 진핵생물 세포보다 원핵생물 세포에서 훨씬 더 빠르게 발생한다. 세균은 때때로 40분밖에 걸리지 않지만, 동물 세포는 최대 400시간이 걸릴 수 있다. 진핵생물은 또한 마지막 염색체 끝에서 텔로미어를 복제하기 위한 뚜렷한 작동 방식을 가지고 있다. 원핵생물은 환형 염색체를 가지고 있어, 합성할 끝이 없다. 원핵생물은 지속적으로 발생하는 짧은 복제 과정을 가지고 있으며, 반면에 진핵생물 세포는 세포 주기의 S-기 동안에만 DNA 복제를 수행한다.
6. 3. 복제 속도
원핵생물과 진핵생물 모두 오카자키 절편을 갖는다.[23] 원핵생물과 진핵생물의 DNA 복제 속도는 다르다. 원핵생물은 진핵생물보다 복제 속도가 빠르다. 세균은 DNA 복제에 약 40분이 걸리지만, 동물 세포는 최대 400시간까지 걸릴 수 있다.원핵생물 세포는 구조가 단순하고, 핵과 세포 소기관이 없으며, DNA는 단일 염색체 형태이다. 반면 진핵생물 세포는 여러 소기관을 가진 핵과 선형 염색체로 배열된 더 많은 DNA를 가지고 있다. 평균적으로 진핵생물 세포는 원핵생물 세포보다 약 25배 많은 DNA를 갖는다.
진핵생물은 염색체 끝에서 텔로미어를 복제하는 특별한 방식을 가지고 있다. 원핵생물은 환형 염색체를 가지므로 복제할 끝이 없다. 원핵생물은 지속적으로 짧은 복제 과정을 거치지만, 진핵생물 세포는 세포 주기의 S-기 동안에만 DNA 복제를 수행한다.
두 생물의 복제 단계는 유사하다. DNA 헬리케이스가 DNA를 풀고, DNA 중합효소가 새로운 가닥을 만든다. 반보존적 복제 패턴에 따라 DNA의 개별 가닥이 다른 방향으로 생성되어 선도 가닥과 지연 가닥이 생긴다. 지연 가닥은 오카자키 절편으로 합성된 후 연결된다. 두 생물 모두 작은 RNA 가닥으로 새로운 DNA 가닥을 시작한다.
6. 4. 텔로미어 복제 (Telomere replication)
오카자키 절편은 원핵생물과 진핵생물 모두에 존재한다.[23] 진핵생물 DNA 분자는 원핵생물의 환형 분자와 달리 더 크고 일반적으로 여러 복제 기원을 갖는다. 이는 각 진핵생물 염색체가 여러 복제 기원을 가진 많은 DNA 복제 단위로 구성됨을 의미한다. 진핵생물에서 이러한 복제 분기점은 DNA를 따라 수없이 많으며 복제 중 DNA에 "버블"을 형성한다. 복제 분기점은 자율 복제 서열(ARS)이라고 하는 특정 지점에서 형성된다. 진핵생물은 클램프 로더 복합체와 증식 세포 핵 항원이라고 하는 6개의 단위 클램프를 가지고 있다.[24] 복제 분기점의 효율적인 이동은 ATP 의존적 클램프 로더 복합체에 의해 지연 DNA 가닥의 새로 프라이밍된 부위에 슬라이딩 클램프를 신속하게 배치하는 데 크게 의존한다. 이는 오카자키 절편의 조각별 생성이 선도 가닥에서 DNA의 연속적인 합성을 따라갈 수 있음을 의미한다. 이러한 클램프 로더 복합체는 모든 진핵생물의 특징이다.[25]진핵생물은 또한 마지막 염색체 끝에서 텔로미어를 복제하기 위한 뚜렷한 작동 방식을 가지고 있다. 진핵생물 세포는 세포 주기의 S-기 동안에만 DNA 복제를 수행한다.
7. 기술적 응용 (Uses in technology)
오카자키 절편 관련 연구는 의학 및 생명공학 분야에 응용될 수 있다.
7. 1. 오카자키 절편 관련 의학적 개념 (Medical concepts associated with Okazaki fragments)
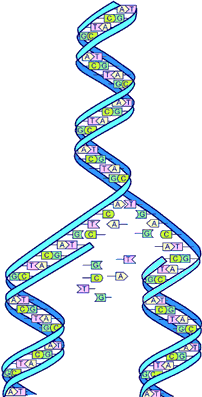
세포는 유전 서열에 돌연변이가 없도록 여러 단계를 거치지만, 때로는 오카자키 절편 성숙 과정에서 특정 결실 및 기타 유전적 변화가 감지되지 않고 지나가기도 한다. 오카자키 절편은 지연 가닥의 뉴클레오타이드 집합이므로, 원래 가닥의 결실, 삽입 또는 중복을 포함한 모든 변화는 감지 및 수정되지 않으면 돌연변이를 일으킬 수 있다.[26] DNA 복제를 돕는 단백질의 문제 또한 돌연변이의 원인이 될 수 있다. 예를 들어, 프리마아제와 관련된 돌연변이는 RNA 프라이머 제거에 영향을 미쳐 DNA 가닥을 더 깨지기 쉽게 만들고 파손되기 쉽게 할 수 있다. 폴리머라아제 α 관련 돌연변이는 오카자키 절편 서열 편집과 단백질의 유전 물질 내 삽입을 손상시킨다. 이 두 가지 변화는 모두 염색체 이상, 의도하지 않은 유전자 재배열, 그리고 나중에 다양한 암을 유발할 수 있다.[26]
단백질 돌연변이가 생물체에 미치는 영향을 알아보기 위해 연구자들은 DNA 복제 관련 단백질인 플랩 엔도뉴클레아제 1(FEN1)의 বিভিন্ন 돌연변이에 대해 동형 접합성이 되도록 실험용 쥐를 유전적으로 변형했다. 결과는 특정 유전자 변화에 따라 달랐다. 동형 접합체 녹아웃 돌연변이 쥐는 "세포 증식 실패"와 "초기 배아 치사"를 경험했다. F343A 및 F344A (FFAA라고도 함) 돌연변이가 있는 쥐는 범혈구 감소증 및 폐 폐형성 부전을 포함한 출산 합병증으로 인해 출생 직후 사망했다. 이는 FFAA 돌연변이가 FEN1이 PCNA (증식 세포 핵 항원)와 상호 작용하는 것을 방해하여 오카자키 절편 성숙 과정에서 제 기능을 완료하지 못하게 하기 때문이다. 이 단백질과의 상호 작용은 FEN1의 생물학적 기능에서 핵심적인 분자 기능으로 간주된다. FFAA 돌연변이는 RNA 프라이머 제거 및 긴 염기쌍 복구에 결함을 일으켜 DNA에 많은 파손을 유발한다. 면밀한 관찰 결과, FFAA FEN1 돌연변이에 대해 동형 접합체인 세포는 성숙 과정에서 부분적인 결함만을 나타내는 것으로 보였다. 이는 돌연변이에 대해 이형 접합체인 쥐는 게놈에 여러 개의 작은 틈이 있음에도 불구하고 성년까지 생존할 수 있음을 의미한다. 그러나 이러한 틈은 필연적으로 향후 DNA 복제를 방해한다. 파손으로 인해 복제 포크가 붕괴되고 실제 DNA 서열에 이중 가닥 파손이 발생하기 때문이다. 시간이 지남에 따라 이러한 틈은 전체 염색체 파손을 유발하여 심각한 돌연변이와 암으로 이어질 수 있다. 폴리머라아제 α의 변형된 버전으로 구현된 다른 돌연변이도 유사한 결과를 초래했다.[26]
7. 2. 유전 질환 연구 및 치료제 개발
세포는 유전 서열에 돌연변이가 없도록 여러 단계를 거치지만, 때로는 오카자키 절편 성숙 과정에서 특정 결실 및 기타 유전적 변화가 감지되지 않고 지나가기도 한다. 프리마아제와 관련된 돌연변이는 RNA 프라이머 제거에 영향을 미쳐 DNA 가닥을 더 깨지기 쉽게 만들 수 있다. 또 다른 돌연변이는 폴리머라아제 α에 관한 것으로, 오카자키 절편 서열 편집과 단백질의 유전 물질 내 삽입을 손상시킨다. 두 가지 변화 모두 염색체 이상, 의도하지 않은 유전자 재배열, 그리고 나중에 다양한 암을 유발할 수 있다.[26]DNA 복제와 관련된 단백질인 플랩 엔도뉴클레아제 1(FEN1)의 돌연변이를 연구하기 위해 실험용 쥐를 유전적으로 변형한 실험이 진행되었다. 동형 접합체 녹아웃 돌연변이 쥐는 세포 증식 실패와 초기 배아 치사를 경험했고, F343A 및 F344A (FFAA) 돌연변이를 가진 쥐는 범혈구 감소증 및 폐 폐형성 부전을 포함한 출산 합병증으로 인해 출생 직후 사망했다. FFAA 돌연변이는 FEN1이 PCNA(증식 세포 핵 항원)와 상호 작용하는 것을 방해하여 오카자키 절편 성숙 과정에서 기능을 완료하지 못하게 한다. 이로 인해 RNA 프라이머 제거 및 긴 염기쌍 복구에 결함이 발생하고, DNA에 많은 파손이 발생한다. 이러한 틈은 향후 DNA 복제를 방해하고, 파손으로 인해 복제 포크가 붕괴되고 실제 DNA 서열에 이중 가닥 파손이 발생한다. 시간이 지남에 따라 이러한 틈은 전체 염색체 파손을 유발하여 심각한 돌연변이와 암으로 이어질 수 있다.[26]
참조
[1]
논문
Okazaki fragment metabolism
2013-02
[2]
논문
Days weaving the lagging strand synthesis of DNA - A personal recollection of the discovery of Okazaki fragments and studies on discontinuous replication mechanism
2017-05-11
[3]
서적
The Cell: A Molecular Approach
Sinauer Associates
2000
[4]
논문
DNA replication: partners in the Okazaki two-step
2001-10
[5]
논문
Discontinuous DNA replication
[6]
논문
Mechanism of DNA chain growth. I. Possible discontinuity and unusual secondary structure of newly synthesized chains
1968-02
[7]
논문
Mechanism of DNA chain growth, II. Accumulation of newly synthesized short chains in E. coli infected with ligase-defective T4 phages
1968-08
[8]
논문
Phosphate steering by Flap Endonuclease 1 promotes 5′-flap specificity and incision to prevent genome instability
https://www.nature.c[...]
2024-11-05
[9]
논문
An alternative pathway for Okazaki fragment processing: resolution of fold-back flaps by Pif1 helicase
2010-12
[10]
논문
DNA primase acts as a molecular brake in DNA replication
https://pure.rug.nl/[...]
2006-02
[11]
논문
DNA ligase I deficiency leads to replication-dependent DNA damage and impacts cell morphology without blocking cell cycle progression
2009-04
[12]
논문
Okazaki fragment maturation in yeast. II. Cooperation between the polymerase and 3'-5'-exonuclease activities of Pol delta in the creation of a ligatable nick
2003-01
[13]
논문
An interaction between DNA ligase I and proliferating cell nuclear antigen: implications for Okazaki fragment synthesis and joining
1997-11
[14]
논문
Interaction between PCNA and DNA ligase I is critical for joining of Okazaki fragments and long-patch base-excision repair
[15]
논문
The 3'→5' exonuclease of DNA polymerase delta can substitute for the 5' flap endonuclease Rad27/Fen1 in processing Okazaki fragments and preventing genome instability
2001-04
[16]
논문
Flap endonuclease 1: a central component of DNA metabolism
[17]
논문
Coupling of DNA helicase and endonuclease activities of yeast Dna2 facilitates Okazaki fragment processing
2002-07
[18]
논문
Characterization of the enzymatic properties of the yeast dna2 Helicase/endonuclease suggests a new model for Okazaki fragment processing
2000-12
[19]
논문
Dna2 on the road to Okazaki fragment processing and genome stability in eukaryotes
2010-04
[20]
논문
Significance of the dissociation of Dna2 by flap endonuclease 1 to Okazaki fragment processing in Saccharomyces cerevisiae
2009-03
[21]
논문
Okazaki fragment processing-independent role for human Dna2 enzyme during DNA replication
2012-06
[22]
논문
Okazaki fragment maturation in yeast. I. Distribution of functions between FEN1 AND DNA2
2003-01
[23]
웹사이트
Are Okazaki fragments unique to eukaryotes? Or is it universal, so it's present in bacterial DNA replication as well?
https://www.life.ill[...]
School of Molecular and Cellular Biology, University of Illinois at Urbana-Champaign
[24]
웹사이트
Eukaryotic DNA Replication.
http://molecular-pla[...]
multilab.biz
2011-03-29
[25]
논문
Identification of short 'eukaryotic' Okazaki fragments synthesized from a prokaryotic replication origin
2003-02
[26]
논문
Okazaki fragment maturation: nucleases take centre stage
2011-02
[27]
웹사이트
岡崎フラグメントと私
https://brh.co.jp/s_[...]
生命誌研究会
2021-11-13
[28]
논문
Discontinuous DNA transcription
1980
[29]
논문
Mechanism of DNA chain growth. I. Possible discontinuity and unusual secondary structure of newly synthesized chains
1968-02
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com
